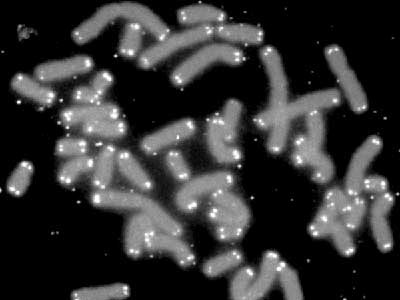
 Szóval, miről is van szó. A telomérák az eukarióta kromoszómák végén levő rövid, ismétlődő szakaszok, amelyek a kromoszómán levő DNS fennmaradó részét védik az élőlény élete során.
Szóval, miről is van szó. A telomérák az eukarióta kromoszómák végén levő rövid, ismétlődő szakaszok, amelyek a kromoszómán levő DNS fennmaradó részét védik az élőlény élete során.
Először Elizabeth Blackburn fedezte fel őket, egy csillós egysejtűt, a Tetrahymenát vizsgálva. Pontosabban, akkor még nem tudták mit is fedeztek fel, csak annyi látszott, hogy a kormoszómák végén rövid, repetitív szekvenciák vannak, amelyek funkciója teljesen misztikus volt.
A kulcskísérlethez a löketet egy konferencia hozta, ahol Blackburn megismerte Jack Szostak munkáját. Szostak ekkortájt mesterséges kromoszómákkal foglalkozott, amelyeket élesztő sejtekbe vitt be és figyelte, hogy mennyiben viselkednek különbözően az élesztő saját kromoszómáitól. Bosszúságára folyamatosan azzal kellett szembesülni, hogy a mesterséges kromoszómák populációs szinten instabilnak bizonyultak, és néhány generáció után egyszerűen eltűntek. Blackburnnel közösen egy triviális kísérletre vállalkoztak: megnézni, hogy mi lesz, ha a Tetrahymena ismétlődő szekvenciáit ráteszik a mesterséges kromoszómák végére. A mai tudásunk számára nem túl meglepő, amit láttak: a kromoszómák éltek és virultak.
Később Blackburn egyik poszt-dokja, a díj harmadát kapó Carol Greider, azt is bebizonyította, hogy a furcsa kis ismétlődéseket egy enzim, a telomeráz hozza létre.
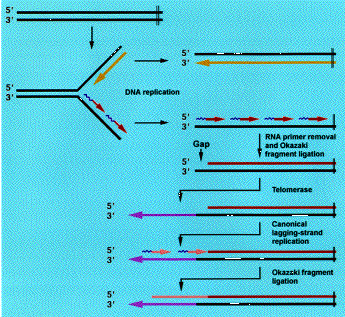 De mire való ez a furcsa rendszer? Az egészre az eukarióta sejtek sajátosságai, és a DNS másolás sutasága miatt van szükség. Ez a sutaság abból ered, hogy a DNS polimerázok csak 5'-3' irányban képesek dolgozni.
De mire való ez a furcsa rendszer? Az egészre az eukarióta sejtek sajátosságai, és a DNS másolás sutasága miatt van szükség. Ez a sutaság abból ered, hogy a DNS polimerázok csak 5'-3' irányban képesek dolgozni.
Amikor a replikációs villa elindul ez azt is jelenti, hogy az egyik szálon gyakorlatilag végig tud futni ugyanaz a polimeráz (ez az ún "leading strand"), mígy a szembenlevő, komplementer szálon ('lagging strand'), ahogy az kinyílik folyamatosan kisebb replikációs egységek jelennek meg. Ezek az Okazaki fragmentumok. (Itt egy részletesebb leírás a DNS replikációról, a PTE honlapján.)
A DNS polimerázok másik jellegzetessége, hogy nem tudnak csak úgy belecsapni a lecsóba, s csak akkor képesek elindítani a szintézist, ha már van mihez kapcsolniuk a nukleotidokat, vagyis szükség van egy primerre. Ezek apró RNS darabok lesznek, amelyek aztán később elemésztésre kerülnek. Így persze az Okazaki fragmentumok közt apró rések keletkeznek, de ezeket már könnyű betömni, hiszen az előző fragmentum szolgál majd primerként a polimeráz számára. Egy kivétel van, az utolsó (pontosabban legelső), az 5' végen levő primer. Ezt nem lehet pótolni, mert előtte már nincs senki. Így amikor ez elemésztődik, egy egyszálú templát-DNS darab fog kilógni a kromoszóma végén. Ugyanez igaz a leading strandre is, hiszen ez is egy RNS primerről indul, és az általa lefedett szekvenciarész nem lesz pótolható.
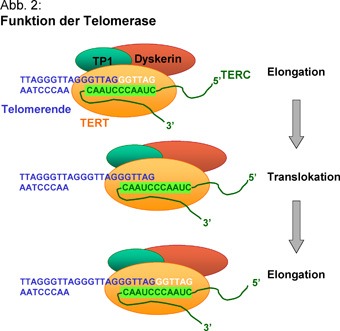 A sejtekben a polimerázok mellett nukleázok is találhatók, olyan enzimek, amelyek DNS-t emésztenek, különösen akkor, ha az egyszálú és a vége a semmibe lóg. (Megj: baktériumok esetében a DNS körkörös plazmidokon helyezkedik el, ott nem is létezik ez az egész probléma.) Azaz minden egyes sejtosztódáskor a kormoszómák végéből lecsípődne egy darab. Ez nem lenne túlzottan adaptív, és erre jelentenek megoldást a telomérák és a telomeráz enzim.
A sejtekben a polimerázok mellett nukleázok is találhatók, olyan enzimek, amelyek DNS-t emésztenek, különösen akkor, ha az egyszálú és a vége a semmibe lóg. (Megj: baktériumok esetében a DNS körkörös plazmidokon helyezkedik el, ott nem is létezik ez az egész probléma.) Azaz minden egyes sejtosztódáskor a kormoszómák végéből lecsípődne egy darab. Ez nem lenne túlzottan adaptív, és erre jelentenek megoldást a telomérák és a telomeráz enzim.
A telomeráz enzim egy apró RNS templátot felhasználva, gyorsan képes újakat szintetizálni a telomérákból, ami a kormoszóma meghosszabbodásához vezet, illetve friss replikáció esetén, a leemésztődő szekvenciák pótlására szolgál. Azaz a rendszer lehetővé teszi, hogy az eukarióta replikáció egyáltalán működjön, folyamatos genetikai anyag elvesztegetés nélkül.
Ha a rendszer elromlik (túl aktívvá válik, vagy belassul) az gondot jelent. A rák és az öregedés esetében figyelhetünk meg ilyesmit.



Utolsó kommentek