A kétoldali szimmetriájú állatok két nagy csoportra oszlanak, aszerint, hogy az ősbélüreg (archenteron) nyílásából a szájnyílás vagy a végbélnyílás alakul ki. Az előbbi csoportot Protostomiának (ez többé kevésbé megegyezik az "ősszájúak" fogalmával), míg utóbbit Deuterostomiának ("újszájúak") nevezzük. (Van itt a levegőben néhány örökérvényű poén, hogy a két csoport kölcsönösen minek tarthatja egymást, de most inkább nem lőném le őket... ;-)) Az eddigi genomprojectek adatainak egyik érdekes hozadéka, hogy a Protostomia csoportba tartozó rovarok (Drosophila, háziméh) és fonalférgek (C. elegans) genomja kompaktabb és kevesebb intront tartalmaz mint a Deuterostomia csoporthoz tartozó a gerinces genomok. (Az intronok olyan, fehérjét nem kódoló szekvenciák, melyek a genomi DNS-ben a kódoló szekvenciák (exonok) közé ékelődve vannak jelen, azonban az mRNS transzkripciója közben kivágódnak a végső "termékből", így a fehérje szintézis helyszínére már csak a kódoló szekvenica kerül.) Erre két magyarázat létezhet: a valamikori közös ősben (Urbilateria) kevés intron volt és a gerincesek vonala szedett össze újakat, vagy a közös ősben sok intron volt, de a rovarok valahogy elvesztették őket.
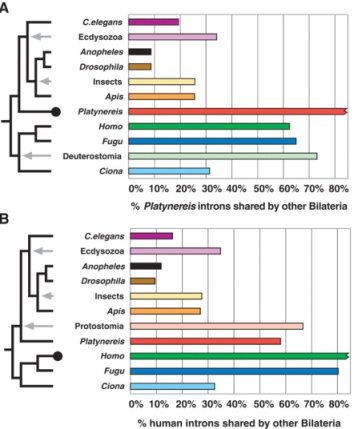 1. Ábra: A Platynereis intronjainak helyét összevetve más
állatok intronjaival, a legnagyobb megegyezést a gerincesekkel találjuk
(A). Hasonlóan, az emberi intronok helyzetét összevetve más állatokéval
a Platynereis genomja kiugróan magas hasonlóságot mutat a
Protostomia csoportban (B). (A szürke nyilakhoz tartozó csoportnevek
hasonlósági értékei azért magasabbak mint a csoport egyes tagjaiéi,
mert a csoportban fellelhető összes intront ábrázolják - vagyis durván
extrapolálnak a közös ős genomjára.)
1. Ábra: A Platynereis intronjainak helyét összevetve más
állatok intronjaival, a legnagyobb megegyezést a gerincesekkel találjuk
(A). Hasonlóan, az emberi intronok helyzetét összevetve más állatokéval
a Platynereis genomja kiugróan magas hasonlóságot mutat a
Protostomia csoportban (B). (A szürke nyilakhoz tartozó csoportnevek
hasonlósági értékei azért magasabbak mint a csoport egyes tagjaiéi,
mert a csoportban fellelhető összes intront ábrázolják - vagyis durván
extrapolálnak a közös ős genomjára.)
Az aktuális Science egyik cikke a vitát az utóbbi magyarázat javára látszik eldönteni [1]. Egy tengeri gyűrűsféreg, a Platynereis dumerilii exon-intron szerkezetét vizsgálva kiderült, hogy bár a Protostomia csoporthoz tartozik, az intronok géneken belüli helyzete és átlagos száma (7.8) sokkal közelebb van a gerincesekéhez (8.4), mint a Protostomián belüli Ecdysozoa csoportban levő rovarokéhoz és fonalférgekéhez (2.4-5.4). (1. Ábra) Gyorsan megjegyezném, hogy ez nem azt jelenti, hogy az eddigi filogenetikai fák fabatkát sem érnek. Ezeket az orthológ szekvenciák összehasonlítása alapján készítették és igen megbízhatók. Itt mindössze intronok helyéről van szó és nem a szekvenciájukról. Így a Platynereis megmarad ősszájúnak, mégha az ebből a csoportból vizsgált fajok közül filogenetikailag is ő áll hozzánk a legközelebb.
A cikkből levonható legfontosabb következtetés az, hogy a közös ősnek, az Urbilateriának, minden valószínűségel a gerincesekéhez nagyon hasonló exon-intron szerkezete lehetett, s míg a rovarok evolúciója során a genom igen gyorsan változott és kompaktabb lett, addig ez bennünk és a tengeri gyűrűsférgekben ez nem következett be. Mi - ebből a szempotból - kicsit lassúak voltunk és ezért egy primitívebb állapotot tükrözünk ;-). A szerzők szavaival:
Platynereis and humans can be regarded as similarly slow-evolving representatives of protostomes and deuterostomes, respectively.Az eredmény érdekes és természetesen kicsit meglepő, ugyanakkor nem teljesen váratlan, mert már voltak jelek a komplexebb, nagyobb genom ősibb jellegére- és a kompakt genomok későbbi kialakulására vonatkozóan. Az egyik ilyen jel, hogy bár a gerincesekben fellelhető Wnt fehérje 12 alcsaládjából 11 szintén megtalálható egy csalánozó fajban (a csalánozók - pl. hidrák - körkörös szimmetriájúak, vagyis a csalánozók és a kétoldali szimmetriájú állatok közös őse az Urbilateria előtt létezett), a Nematostella vectensis-ben, a rovarokban csak hat alcsalád lelhető fel, azaz néhány elvesztődött a hosszú évek alatt [2]. Ráadásul úgy tűnik, hogy a Drosophila olyannyira előrehaladt a nem létfontosságú DNS szekvenciák kiszűrésében, hogy minden ami a genomjában maradt az szigorú szelekció alatt van [3], és valójában szinte egyáltalán nincs fölösleges, ún. "szemét DNS-e" ("junk DNA"), ami a neutrális evolúció fontos játéktere és -szere lenne. Na, de erről majd valamikor máskor. ;-)
[1] Raible F, Tessmar-Raible K, Osoegawa K, Wincker P, Jubin C, Balavoine G, Ferrier D, Benes V, de Jong P, Weissenbach J, Bork P, Arendt D (2005) Vertebrate-type intron-rich genes in the marine annelid Platynereis dumerilii. Science 310: 1325-1326.
[2] Miller DJ, Ball EE, Technau U. (2005) Cnidarians and ancestral genetic complexity in the animal kingdom. Trends Genet. 21(10): 536-539.
[3] Andolfatto P. (2005) Adaptive evolution of non-coding DNA in Drosophila. Nature 437: 1149-1152.



Utolsó kommentek